科技日报记者 张梦然
在新一期《科学》期刊上,诺贝尔奖得主、美国华盛顿大学的大卫·贝克及其团队发表了一篇突破性研究论文:他们首次利用人工智能(AI)技术,从零开始设计了具有复杂活性位点的丝氨酸水解酶。这项成就标志着酶工程领域的一个重要里程碑,表明现在人们有能力设计出具有天然酶活性的酶,并且这些人工设计的酶还具备实际应用潜力。
传统方法在设计能够催化特定化学反应的酶时,面临巨大挑战,主要是因为将活性位点整合到预先存在的蛋白质结构中时,往往会受到结构灵活性和活性位点预组织程度的限制,从而影响催化效率。尽管化学手段可以在一定程度上克服这些问题,但最初计算设计的酶效率仍远低于天然酶。
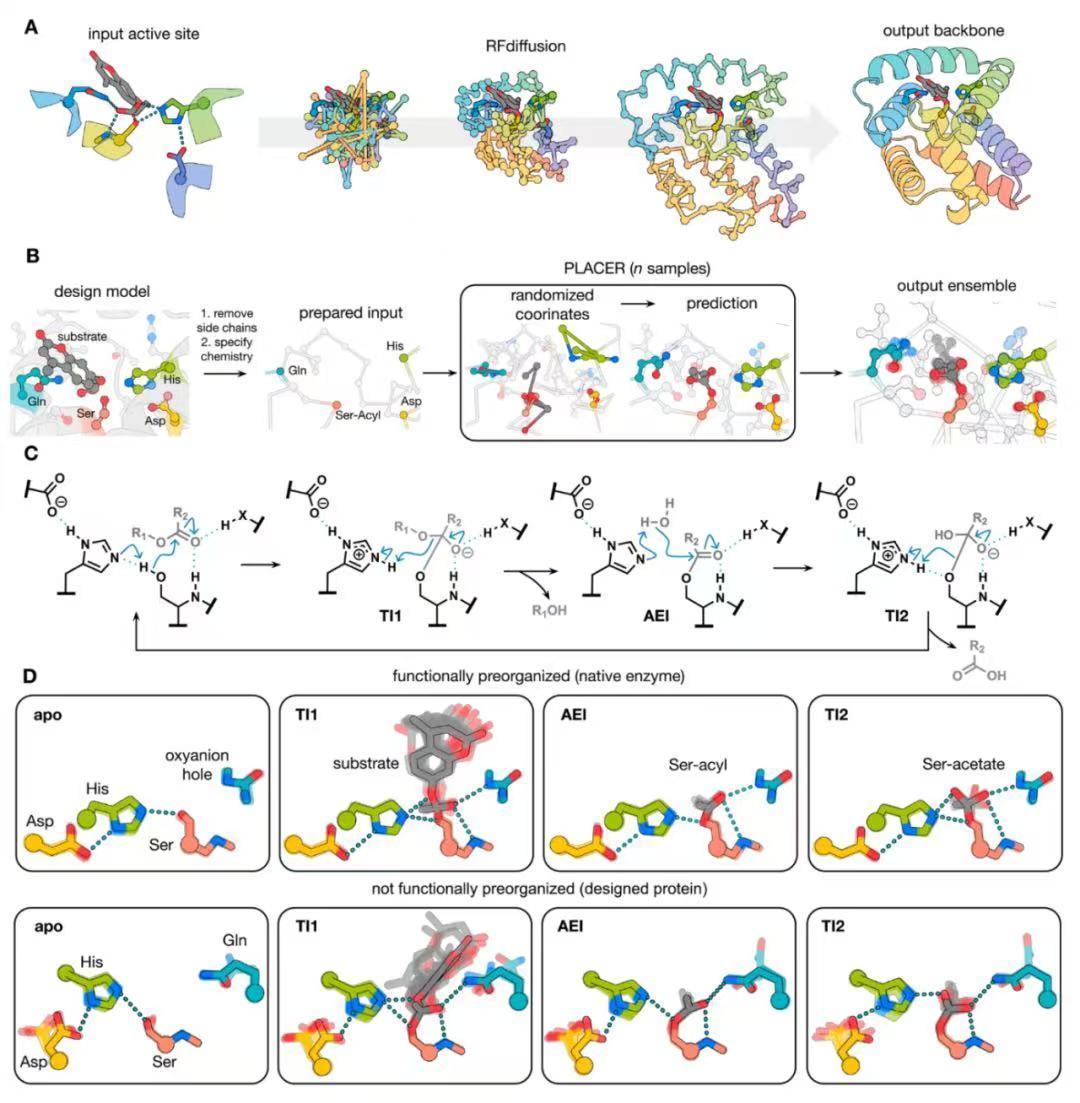
然而,随着深度学习技术的发展,为从头设计蛋白质提供了可能,尤其是对于那些拥有复杂活性位点的蛋白质,如丝氨酸水解酶——这是已知最大的一类酶家族之一。贝克团队此次引入了一种名为PLACER(蛋白质—配体原子构象集合再现)的新型机器学习网络,该网络通过分析蛋白质骨架、氨基酸特性和结合分子的化学结构,来预测酶活性位点的精确原子排列。
团队采用了名为RFdiffusion的技术来创建含有复杂催化位点的蛋白质,并使用PLACER评估这些蛋白质的活性位点组织情况。最终,他们成功设计出了功能性丝氨酸水解酶,这些酶仅需最小化的活性位点规范就能有效地催化酯水解反应。此外,这种方法还在低通量筛选过程中发现了5种全新的酶折叠方式,这些方式不同于任何已知的天然丝氨酸水解酶中的折叠,极大地扩展了这一古老酶家族的结构多样性。
这项研究不仅代表了从头设计酶的重大进步,也为开发更高效的催化剂开辟了新途径,同时对理解和扩展酶的功能性具有重要意义。
总编辑圈点
在生命科学领域,人工智能正为蛋白酶设计带来变革性突破。蛋白酶的设计通常需要精确控制氨基酸序列和三维结构。传统的蛋白酶设计依赖大量的实验试错,既耗时又成本高昂。而人工智能凭借强大的算法和数据处理能力,能够快速分析海量的蛋白质结构与功能数据,并预测氨基酸序列如何折叠成三维结构。人工智能设计蛋白酶不仅显著提升了设计效率,而且有助于发现和设计全新的蛋白酶,为生命科学领域的研发创新注入新活力。